育种专栏 | 7-基因分型之靶向测序
前沿分享
2022-11-04
测序技术发展至今,其应用向两个极端方向发展,一是大而全的全基因组测序,二是小而精的靶向测序。
靶向测序简介
靶向测序是将一组目标基因或基因组区域分离出来,并进行富集和测序的方法。该方法使得研究人员将时间、费用和数据分析聚焦在感兴趣的特定区域(靶标区域、基因),利用较少的数据量就能得到较高的灵敏度和准确度,实现对变异位点的快速筛选。这些靶标区域通常包括外显子组(基因组的蛋白质编码部分)、感兴趣的特定基因(定制内容),以及基因或线粒体DNA中的目标区域等。
相比于全基因组测序(WGS)和全外显子测序(WES),靶向测序能够去除冗余数据的干扰,使用更低成本和更高深度的方式,最大限度地利用测序reads,尤其适用于在临床应用中样本量少时的情况。比如遗传突变、肿瘤筛查等领域,在分析指定样本的特定突变时,靶向的基因测序面板(panel)是非常有用的工具。重点panel包含了一组精选的基因或基因区域,它们与正在研究的疾病或表型有着已知或疑似的关联。在定制基因panel时,可挑选预先选定的内容,或定制设计内容,从而将感兴趣的基因组区域包含在内,如此就可以节省资源并最大程度地减少数据分析的工作量。
靶向测序在肿瘤基因检测、遗传病基因检测、病原微生物检测等领域已经广泛应用,但在农业育种上的应用仍亦步亦趋,其原因小编认为主要有三:一是动植物基础研究不够,数据量和已知位点太少;二是方法和技术上的适用性和创新性不足,只能玩人类研究剩下的;三是动植物个体价值太低,育种成本较高。
但是,经过科研人员这么多年的努力,基于靶向测序的分子育种技术和平台已经开始成型,各家检测公司也推出各式各样的产品(但原理基本都一样),并逐渐发挥出它的巨大应用价值,如更加精准和快速定位与特定性状相关的候选基因,进行动/植物品系鉴定,优势性状筛选,辅助育种分子标记开发,群体遗传学分析以及种质亲缘关系分析等。
靶向测序原理
从技术原理上,靶向测序可分为杂交捕获测序和多重扩增子测序两种。
液相杂交捕获测序
杂交捕获测序是指通过设计与目标片段互补的生物素化探针,使其与含目的基因的片段进行杂交,将目的基因片段富集后进行高通量测序。根据支持物的不同,探针杂交捕获技术分为液相杂交与固相杂交。固相杂交由于其在花费与操作上的劣势,已基本被淘汰;液相杂交是在溶液中,目标片段和带有生物素标记的探针直接杂交,然后利用被链霉亲和素包裹的磁珠对杂交到生物素探针的片段进行吸附。洗去游离DNA后,将富集得到的DNA进行扩增,构建高通量测序文库。
液相杂交捕获测序基于碱基互补配对原理,设计合成核酸探针,对DNA文库进行基于液相环境的目标区域杂交富集后测序,它适用于几kb到上百Mb的基因组目标区域的检测,可检测SNV、InDel、CNV、SV和基因融合等变异。

多重PCR扩增子测序
多重扩增子测序是针对感兴趣的目标区域,设计多重PCR引物进行扩增富集并进行测序。通常适用于检测几十到几千个位点,或几十kb以下的区域。
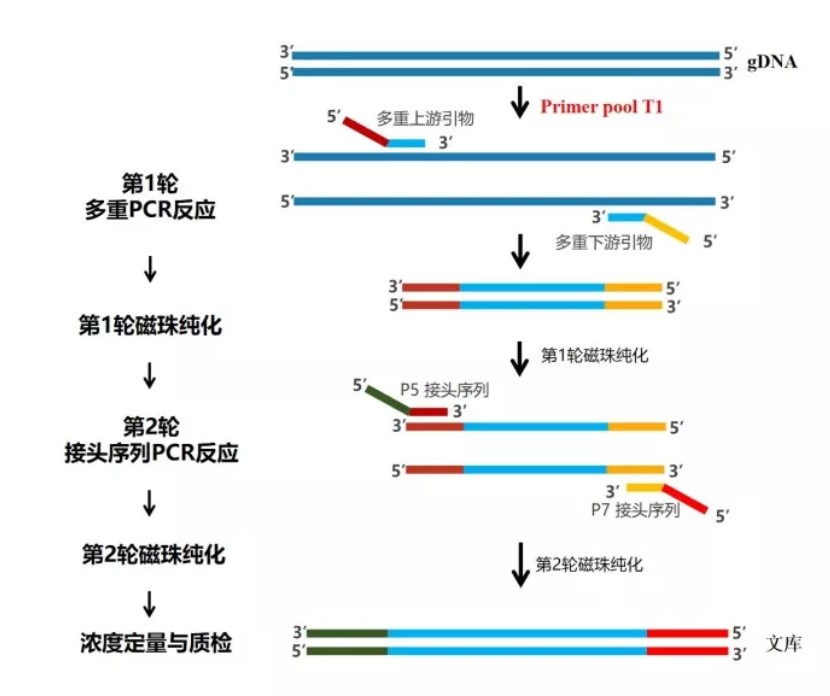
两种靶向测序方法与固相芯片的比较如下表所示:
关于靶向测序在动植物育种中的应用,现在多重PCR已经使用较少,基于液相的杂交捕获相对更常用,不过也要根据具体应用场景而定。一般而言,中高低密度的芯片采用杂交捕获,低密度芯片采用多重扩增子(一般<5K),适用于大样本量的重要位点检测,类似于KASP标记,但比KASP标记还是相对要多一些。
靶向测序数据指标
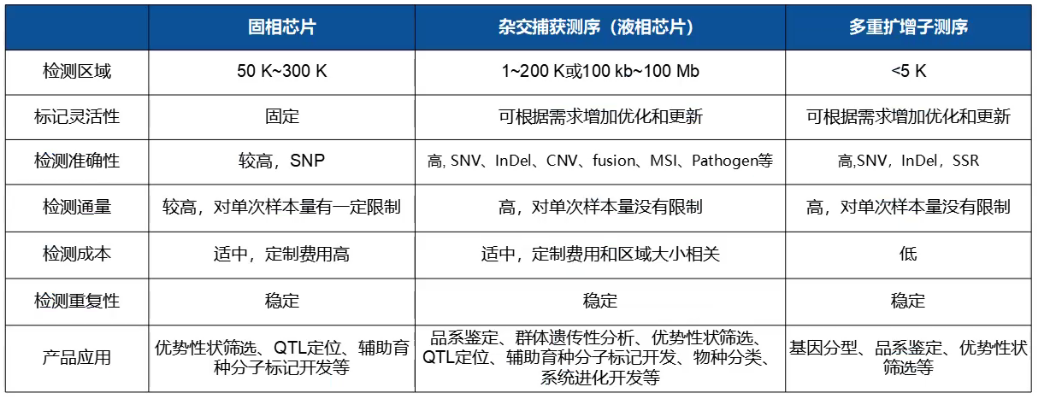
目标区域捕获测序的数据质量主要通过目标区域覆盖度、捕获特异性(捕获效率)、目标区域覆盖均一性等数据指标来评价。目标区域覆盖度是指目标区域能够被测到的比例。最理想情况是目标区域覆盖度100%,但由于探针设计时会考虑GC含量、序列特征和拷贝数等各种因素,有些区域的探针会影响到整个panel的效果,为保证整体的基因捕获效率,会选择放弃大约0.1-3%区域的捕获。由于碱基互补配对会存在一些错配,因此在捕获目标区域的同时,探针也会结合一些序列相似的非目标区域。落在目标区域的数据占总数据的比例,就是捕获特异性(或捕获效率)。捕获效率高,意味着测序数据的利用率高。提升捕获效率的方式有很多,如优化探针的设计方法,改进重复序列封闭组分、接头封闭组分,优化杂交条件包括缓冲液、杂交流程、漂洗严谨性等等。目标区域位点深度的分布符合泊松分布(近似正态分布),我们一般用20%平均深度的覆盖度或50%平均深度的覆盖度来评价数据的均一性。对于均一性好的数据,其深度的分布图会呈现很窄的峰,即50%平均深度的覆盖度会比较高;而均一性不好的数据,其位点深度分布会更离散,呈现出很宽的峰,就会有很大比例的位点覆盖深度是低于50%平均深度的。
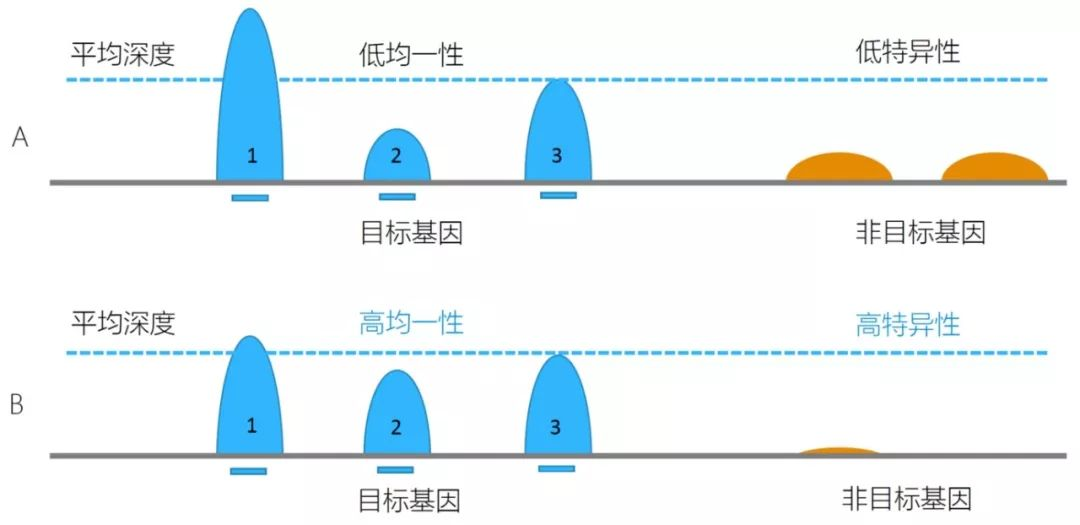
此外,与重测序数据结果的一致性也可作为评价指标之一。
MNP(mSNP)标记
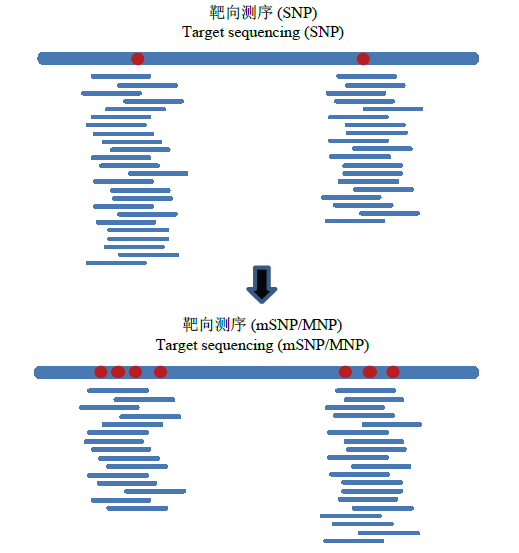
一般而言,每一个SNP 标记设计一对特定的扩增引物,在所获得的扩增子内产生一个SNP 标记,即一个扩增子对应一个SNP 标记。因此,总体来看,所检测到的SNP 在基因组上形成单个的均匀分布。为了最大限度地利用每对引物扩增所获得的DNA片段的信息,由农业农村部科技发展中心与江汉大学合作研发了一种在单个扩增子内可以检测多个SNP,称之为多聚单核苷酸多态性(mSNP 或 MNP)的技术。
与每个扩增位点只包括一个SNP 的液相芯片相比,mSNP 液相芯片具有四方面的改进。一是在每一个扩增位点可以产生多个SNP 标记,即多个SNP的聚合体(mSNP),使可检测的SNP 数目扩大到位点数的4 倍以上。二是同一扩增子内的多个SNP 标记之间可以构成单倍型,提高了变异的检测效率。三是可以从每个mSNP(扩增子)内挑选次等位基因频率(MAF)最大的SNP 组成核心标记。四是mSNP提供了更为精细的遗传变异检测,包括mSNP 位点内和位点间的变异,而且可以采用单倍型和SNP两种方式分别进行检测。mSNP 技术不仅大大提高了标记的利用率,同时通过“一点多标”提升了标记鉴定的准确度和灵敏度。
MNP技术填补了国内实质性派生品种DNA鉴定标准的空白;首次系统分析了我国1万多个(3000多万对)授权水稻、玉米品种的实质性派生关系,有利于掌握我国植物种质创新水平;实现植物品种DNA鉴定技术的国产化。利用上述技术成果,研制了植物品种DNA鉴定国家标准和技术规范,构建了1万多个授权植物品种的DNA指纹数据库,实施了100多万份次的品种选育、授权、打假与维权鉴定。
参考资料
https://www.zhihu.com/question/485449333
https://blog.csdn.net/qq_22253901/article/details/119676933
基因谷:高质量基因捕获测序如何实现?
植物品种鉴定 MNP 标记法,GB/T 38551-2020.
徐云碧 等,靶向测序基因型检测(GBTS)技术及其应用, 中国农业科学2020,53(15):2983-3004.
Guo Z, Yang Q, Huang F, et al. Development of high-resolution multiple-SNP arrays for genetic analyses and molecular breeding through genotyping by target sequencing and liquid chip. Plant Commun. 2021;2(6):100230.
上一篇:
相关推荐
百奥云基于二代/三代测序数据以及中间结果文件(如fq/bam),自研局部组装算法与流程,并结合了大数据产品基因云湖(GenoLake)的海量数据管理、查询与分析等功能,为用户提供个性化的局部组装服务。通过百奥云的局部组装方案,用户可以快速获取基因/蛋白全长序列信息,避免传统实验所带来的时间和资源浪费,加快决策周期,提高研究和生产效率。同时,还可有效地解决大规模测序数据存储、查询和分析等问题,提高数据利用率,获取更全面的基因组信息,助力动植物研究和育种生产的顺利进行。
AI大数据 | 百奥云育种数据科学家顾林林发表一种高效精准的集成学习基因组选择方法ELPGV
是否存在一种方法可以整合各种模型的结果以期望获得更加精准的预测呢?
近日,湖南农业大学刘次桃/段美娟团队在国际著名期刊《Journal of Integrative Plant Biology》上发表研究论文:The OsWRKY72–OsAAT30/OsGSTU26 module mediates reactive oxygen species scavenging to drive heterosis for salt tolerance in hybrid rice。百奥云大数据部门负责人彭建祥以共同作者身份为本研究提供了个性化的数据分析支持。
水稻智能育种联盟 | 水稻种质资源、品种观摩暨育种创新提升工程恳谈会圆满举行
2024年3月21日上午,水稻智能育种联盟2024水稻种质资源、品种观摩暨育种创新提升工程恳谈会圆满举行,本次会议共有三十余名水稻种业同仁参会,分别来自十余家种企及科研单位,就如何充分利用数字化能力,将我国的种质资源优势进一步转化为育种创新优势的问题齐聚一起,共商水稻种质资源交流与合作共赢,探索水稻品种自主创新能力提升路径
百奥云本次大会携水稻基因体检服务首次参展,诚邀各位专家、老师及同仁共同探讨探讨未来农业发展的新趋势和技术创新。
会议邀请 | 百奥云与您相约2024中国种子(南繁硅谷)大会
百奥云参加此次大会并在B11展台进行展示,现场还有产品讲解及抽奖小礼品。欢迎广大业界同仁前来展台交流,共谋发展,我们在此真诚期待您的到来。


